Si quieres ser más inteligente, la biblioteca es un buen lugar para empezar. Y para los investigadores del cáncer, la selección de un tratamiento más inteligente para los pacientes puede empezar con una biblioteca de genes de cáncer.
En un estudio publicado este mes en Clinical Cancer Research, una revista de la Asociación Americana para la Investigación del Cáncer, los investigadores de la Universidad de Osaka han desarrollado una técnica para estudiar una biblioteca de genes en el micrófono de laboratorio -en lugar de un gen específico a la vez- para identificar qué genes cancerígenos impulsan cánceres hepáticos específicos.
Hay esperanza de que esto pueda a su vez ser usado para mejorar el pronóstico y guiar la selección del tratamiento para este cáncer mortal.
El carcinoma hepatocelular (CHC), un tipo de cáncer que comienza en el hígado, es difícil de detectar a tiempo y de tratar, lo que lo convierte en una de las principales causas de muerte por cáncer en todo el mundo.
Existen tratamientos para el HCC, pero algunos funcionan mejor que otros para diferentes personas, y es difícil saber cuál elegir.
Pero ahora, investigadores de Japón han desarrollado un método usando ratones de laboratorio que puede indicar qué cánceres de hígado son impulsados por genes específicos de cáncer, y lo que es más, han encontrado un biomarcador que podría señalar a las personas que se beneficiarán más de un tratamiento común de HCC.
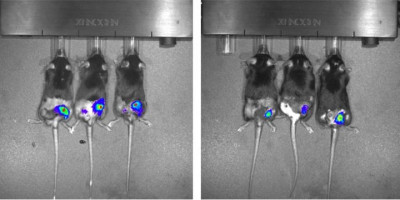
Para ello, los investigadores primero tomaron diez genes de cáncer que se sabe que están involucrados en las vías que conducen al HCC y los usaron para construir una "biblioteca" de ADN inyectándolos en ratones de laboratorio.
Cuando los ratones desarrollaron tumores hepáticos, la secuenciación de los genomas de los tumores mostró qué tumores estaban asociados con qué genes de la biblioteca.
Luego, los ratones con cáncer de hígado fueron tratados con el fármaco quimioterapéutico lenvatinib para indicar qué genes cancerígenos de la biblioteca producen tumores contra los que el tratamiento funciona especialmente bien.
"Nuestros resultados sugieren que el CHC impulsado por el FGF19, que por lo general tiene un mal pronóstico, puede ser susceptible a lenvatinib", dice Takahiro Kodama, autor principal del estudio.
"Pero más allá de ese hallazgo específico, la técnica que utilizamos podría ser usada para evaluar la susceptibilidad de la droga a otros conductores genéticos también".
Saber qué gen impulsa un cáncer es una información poderosa, pero para que sea útil en el mundo real del tratamiento del cáncer, debe haber una forma fácil de identificar cuando los pacientes tienen HCC impulsado por el FGF19.
Trabajando en células hepáticas en el laboratorio, el equipo de investigación identificó seis proteínas cuya expresión disminuyó cuando el gen FGF19 fue desactivado.
A partir de estas seis, el equipo determinó que una proteína conocida como ST6GAL1 era la más estrechamente correlacionada con el FGF19 en el HCC.
Pruebas adicionales de muestras biológicas de pacientes reales con cáncer de hígado confirmaron que los niveles séricos de ST6GAL1 podían utilizarse para identificar a los pacientes con HCC impulsado por el FGF19 con un alto nivel de sensibilidad y especificidad.
"Aunque ya existen algunos modelos de ratones con HCC, nuestro sistema puede utilizarse para estudiar cualquier conjunto de genes, y elimina la necesidad de costosos y largos estudios genéticos realizados uno a uno en ratones preparados individualmente", dice Tetsuo Takehara, autor principal del estudio.
"Este nuevo modelo puede ser una herramienta valiosa para la evaluación preclínica de medicamentos y el aumento de la eficacia de la terapia de medicamentos".
El uso de este modelo para demostrar que el cáncer de hígado impulsado por el FGF19 es susceptible de ser tratado con lenvatinib, y además para señalar una proteína específica que podría ser utilizada como biomarcador, muestra el potencial de esta novedosa técnica.
Fuente: OSAKA UNIVERSITY
La Declaración Mundial sobre el Cáncer reconoce que para lograr reducciones importantes en las muertes prematuras, la educación innovadora y las oportunidades de capacitación para los trabajadores de la salud en todas las disciplinas de control del cáncer deben mejorar significativamente.
ecancer desempeña un papel fundamental en la mejora del acceso a la educación para el profesional médico. Todos los días ayudamos a médicos, enfermeras, pacientes y sus defensores a ampliar sus conocimientos y mejorar la calidad de la atención.
Gracias por tu apoyo.